Глаз человека снабжен двумя типами фоторецепторных клеток — палочками и колбочками, которые получили свое название благодаря форме своих внешних частей, содержащих зрительные пигменты (рис. 4.2).
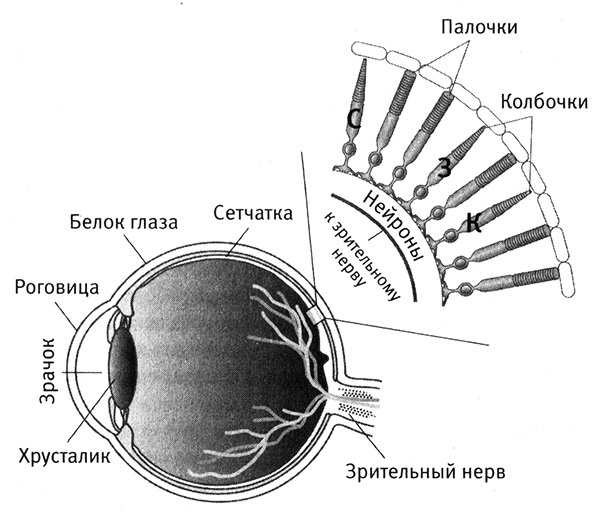
Рис. 4.2. Восприятие цвета сетчаткой человеческого глаза. Свет попадает в глаз через хрусталик, а затем достигает сетчатки. В сетчатке есть два типа фоторецепторных клеток: палочки, которые позволяют видеть при слабом освещении, но не создают цветовых изображений, и колбочки, содержащие опсины одного из трех видов (у человека), настроенные на восприятие красного (К), зеленого (3) или синего (С) цвета. Возбуждение палочек и колбочек интегрируется нейронами, которые соединяются в зрительный нерв. Далее зрительный нерв проводит сигнал к зрительным центрам головного мозга. Рисунок Лианн Олдс.
В целом палочки обладают очень высокой чувствительностью к свету и наиболее интенсивно используются ночью и при слабом освещении, но они не способны различать длину световой волны, поэтому ночью мы плохо различаем цвета. Колбочки используются при ярком свете, и именно с их помощью мы воспринимаем цвет.
Когда свет попадает на хромофор, он вызывает серию очень быстрых изменений зрительного пигмента. За тысячные доли секунды пигмент переходит в возбужденное состояние, что активизирует фоторецепторные клетки. В конечном итоге сигнал фоторецепторов сетчатки достигает зрительных центров коры головного мозга. Чтобы изображение было воспринято как цветное, должны возбудиться как минимум два типа колбочек. Цвет «видимых» мозгом предметов определяется относительным уровнем возбуждения каждого типа колбочек. Если в сетчатке присутствует лишь один тип колбочек, все будет видеться в серой цветовой гамме.
Каждый опсин кодируется отдельным геном. У шимпанзе и других обезьян имеются те же три опсина, что и у человека (SWS, MWS и LWS). Однако у большинства других млекопитающих есть лишь два опсина (и два соответствующих гена), а вот у птиц и рыб — четыре опсина и более. Таким образом, очевидно, что число генов опсинов изменялось в ходе эволюции животных. Эволюция генов опсинов шла через удвоение генов, которое является одним из важных механизмов повышения информационного содержания ДНК. В этом процессе последовательность существующего гена удваивается, а затем пути «старой» и «новой» копии расходятся, и они превращаются в отдельные гены с разными функциями.
Мы знаем, что с опсинами это происходило в ходе эволюции позвоночных животных, но нам хотелось бы знать больше, а именно, какие конкретно животные приобрели (или потеряли) гены опсинов. Недостаточно просто сказать, что два вида различаются между собой: чтобы проследить этапы эволюции, нужно иметь перед глазами картину того, что было до и что было после интересующего нас события. Чтобы узнать истинную историю генов опсинов, нам нужно хорошо представлять себе родственную связь между разными группами животных. Знание родственных связей между организмами позволяет понять направление эволюции конкретного признака и определить состояние этого признака и соответствующего гена у общих предков. Например, если два родственных вида имеют общий признак или общий ген, скорее всего, это не является совпадением, а объясняется тем, что их общий предок также обладал этим признаком или геном. Определить происхождение признака невозможно без знания генеалогии видов. Чтобы представить себе, как была установлена генеалогия видов, имеющих отношение к появлению нашего цветового зрения, нужно хотя бы в общих чертах понять, как сегодня, в эпоху ДНК, устанавливают родственные связи между видами. Я расскажу о недавно обнаруженных и очень важных последовательностях ДНК, которые позволяют реконструировать историю видов с небывалой доселе ясностью и достоверностью.
От молекул к деревьям
С тех пор как Дарвин предположил, что эволюционное развитие организмов можно представить в виде ветвящегося дерева, биологи все время рисуют подобные деревья для изображения родственных связей между ныне живущими и когда-то жившими организмами. Раньше подобные деревья строились на основании внешних признаков современных организмов или ископаемых остатков древних организмов. Однако довольно часто выяснялось, что внешнее сходство или его отсутствие уводит не в ту сторону или по меньшей мере вызывает споры между биологами, поэтому практически для всех основных групп организмов было создано несколько генеалогических деревьев.
Несколько десятилетий назад для определения степени родства между видами ученые начали использовать последовательности биологических молекул. Гены наследуются, и это означает, что последовательности генов и кодируемых ими белков передаются от родителей детям. Происходящие в этих последовательностях изменения также передаются потомкам на всем долгом пути эволюции. Таким образом, степень сходства ДНК может служить показателем близости родства между видами.
Теоретически для построения генеалогического древа биологи имеют в своем арсенале тысячи последовательностей ДНК или белков. На практике для выбора генов для построения древа используют несколько критериев отбора (в частности, стоимость анализа), так что обычно из многих генов-кандидатов выбирают лишь один или несколько. На основании выбранных последовательностей генов с помощью сложного математического и статистического аппарата строят генеалогическое древо. Это древо должно как можно точнее соответствовать имеющимся экспериментальным данным. Так как последовательности генов состоят всего из четырех знаков (A, C, G и T), при недостаточном количестве данных родство между организмами может быть определено неточно. Не следует думать, что с помощью любого гена можно получить один и тот же правильный или однозначный ответ. Поэтому один из способов проверить, насколько верно выстроено древо, — попытаться воспроизвести его на основании другой последовательности или более широкого круга последовательностей.
К счастью, был разработан совершенно новый метод определения родственных связей между видами. Он также основан на анализе ДНК, но учитывает не степень сходства последовательностей в целом, а наличие или отсутствие определенных маркеров в специфических участках ДНК. Эти маркеры возникают в результате случайного встраивания мусорной ДНК рядом с генами. Достаточно часто в ДНК встречаются специфические последовательности, называемые длинными диспергированными повторами (LINE) и короткими диспергированными повторами (SINE). Если такие повторы встраиваются в последовательность, они в ней остаются, поскольку не существует механизма их активного удаления. Последовательность этих элементов в составе ДНК является характерной для данного вида и передается по наследству всем потомкам. Это замечательный инструмент для построения генеалогического древа. Случаи встраивания подобных последовательностей встречаются очень редко, поэтому их присутствие в одном и том же участке ДНК двух разных видов может объясняться исключительно происхождением этих видов от общего предка. На том же принципе наследования маркеров в ДНК основано определение отцовства у людей. Анализ распределения нескольких маркерных элементов ДНК, возникших в разное время у разных организмов, дает биологам достаточно данных, чтобы абсолютно точно устанавливать родственные отношения между видами.